| 미생물의 대사 네트워크 및 유전자조절 네트워크 모델링 기법 개발 | |
|---|---|
| Date 2017-10-09 21:15:36 |
|

교수
건국대학교 KU융합과학기술원 시스템생명공학과
syoon@konkuk.ac.kr
1. 서론
포스트 게놈 시대에 축적된 방대한 데이터와 기술의 진보로 인해 생명현상의 보다 정밀한 측정이 가능해지면서, 생명체를 개별 구성성분이 아닌 전체 시스템 차원에서 분석하고 재해석할 수 있게 되었다. 유전체 수준에서의 유전자 조작이 용이해지면서, 시스템 수준에서의 유기체 조작을 통해 고부가 의약용 단백질이나 바이오 연료 및 유용화학물질 등을 대량생산할 수 있는 생산균주를 제작하려는 시도가 늘고 있다. 이와함께, 새로운 생명체를 제작하거나 기존 생명체를 재설계(re-engineered)하는 과정을 효율적으로 하기 위해 모델링 및 시뮬레이션 기반의 생체 네트워크 설계기술도 크게 주목 받고 있다. 본 기고에서는 생체네트워크의 다양한 수학적 전산적 모델링 기법에 대한 연구동향을 소개한다.
2. 생체네트워크 모델링 기술의 중요성
모델링과 시뮬레이션은 전자공학이나 토목공학에서 이용되는 것처럼 실제로 시스템을 만들기 전에 그 시스템의 거동을 미리 예측하기 위해 이용된다. 비록, 생물시스템의 엄청난 복잡성으로 인해 전기회로 구성이나 교량건축에서처럼 정확할 수는 없지만, 모델링과 시뮬레이션은 미생물과 같은 단일 세포 시스템에 있어서는 부분적으로나마 성공적으로 이용되고 있다. 즉, 생체네트워크의 모델링 및 시뮬레이션을 통해 세포 내 목적생산물 합성경로에 관련된 핵심 조절인자(transcription factor)나 대사회로(metabolic pathway)를 규명할 수 있으며, 생산균주 내에 새로운 유전자회로나 대사회로를 도입하거나 기존 회로를 조작하였을 때 균주 및 생산물의 생산에 미치는 영향을 실험 없이 모사한 후 이를 이용하여 유기체를 우리가 원하는 물질을 대량생산할 수 있는 세포공장(cell factory)으로 개량할 수 있다.
따라서 생체네트워크 모델링 기술은 곧바로 균주개발, 유용물질 생산공정, 생산물 분리, 정제 비용의 감소 등의 전체적인 생물공정 개발에 획기적인 효율성 증대를 가져와 적은 시간, 돈, 인력을 투자하여 더 높은 수준의 생산성을 획득할 수 있게 한다. 특히, 미생물은 그 엄청난 생물다양성(biodiversity)으로 인해 미생물을 이용하여 생산할 수 있는 바이오제품은 무궁무진하다 할 수 있다. 최근의 눈부신 염기서열 해독기법의 발전과 다양한 발현체 분석기법의 출현으로인해 보다 다양한 미생물의 유전체 정보 확보가 가능하며, 이로부터 유용 유전자, 성장, 대사 등에 대한 막대한 양의 정보를 얻을 수 있다. 또한, 미생물의 빠른 증식속도와 균주조작 용이성, 고등유기체에 비해 상대적으로 덜 복잡한 대사및 조절 기작은 유기체의 시스템 수준에서의 통합연구에 적합하다.
3. 대사 네트워크(metabolic network) 모델링
현재까지 개발된 많은 생체반응 모델링 기법 중 실제 생물공정 개발에 이용되는 것은 유전체 수준의 대사 네트워크를 재구성하고 생화학적, 환경적 제약을 부과하여 시뮬레이션을 통해 대사흐름을 분석하는 기법(flux balance analysis, FBA)이다[1].
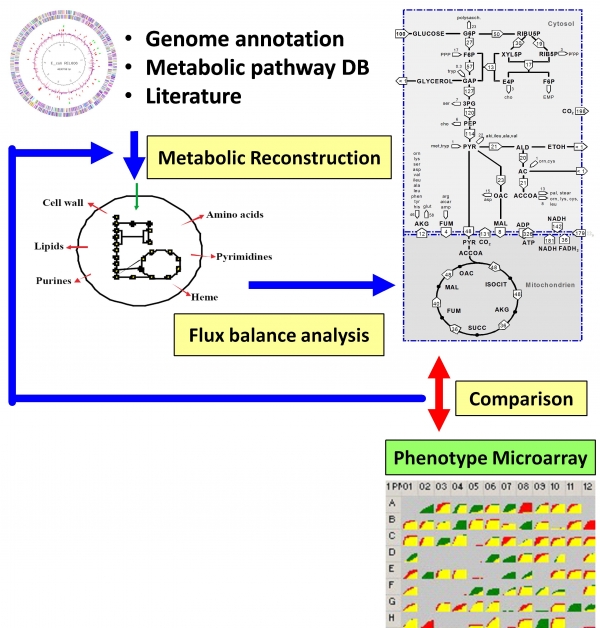
그림 1. 대사 네트워크(metabolic network) 모델링 과정
하지만, 유전체 정보로부터 대사 네트워크를 구성하기 위해서는 해당 균주의 모든 대사반응 정보를 문헌검색을 통해 수작업으로 얻기 때문에 상당한 시간과 노력이 요구된다. 한 유기체의 대사네트워크 모델링의 첫 단계는 유전체 주석(genome annotation) 결과와 Ecocyc, MetaCyc, KEGG와 같은 대사경로 데이터베이스로부터 대사회로 관련 유전자의 기능, 대사 반응(metabolic step), 화학식 등의 정보를 목록화하는 것이다. 대사반응 리스트로부터 초기 대사 모델을 만들고 FBA, gap analysis 등을 통해 필요한 대사반응을 추가하거나 잘못된 대사정보를 수정한다. 만들어진 모델은 Phenotype Microarray와 같은 표현형 데이터와 시뮬레이션 결과를 비교함으로써 그 성능을 검증하고 모델을 보완한다(그림 1). 유전체 해독이 완료된 미생물의 종류가 급증하면서, 최근에는 대사 네트워크 구성작업을 보다 효율적으로 수행하기 위한 자동화 기법이 개발되고 있으며, 대표적으로는 SEED(http://www.theseed.org)를 들 수 있다. 하지만, 많은 연구가 이루어지지 않아 많은 수의 유전자의 기능을 모르거나 새로 발견된 균주의 경우는 대사네트워크를 구성하기 위해서 여전히 많은 시간과 노력이 든다.
4. 유전자 조절 네트워크(gene regulatory network) 모델링
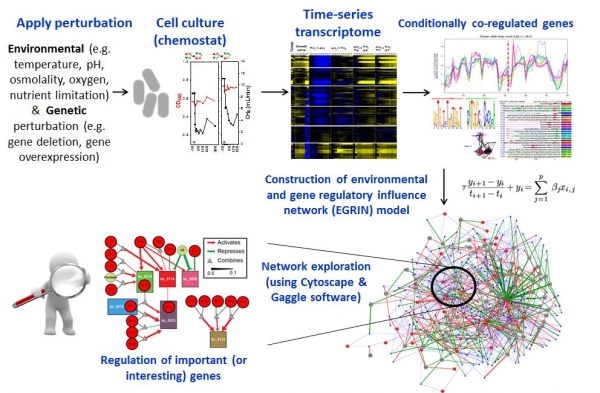
유전자 조절 네트워크(gene regulatory network, GRN)는 시공간적으로(spatiotemporally) 세포내 생리작용(cellular physiology)을 조절하여 자원 활용을 최적화하고, 유전정보를 통합하여 다양한 환경 변화에 유기체가 적응하여 살아갈 수 있도록 해 준다. 거의 모든 유기체는 외부환경과 끊임없이 반응하고 소통하기 위해 조절네트워크가 환경에 따라 변화하기 때문에, 유기체의 조절네트워크의 특성 및 거동 예측은 균주개량에 매우 유용하게 이용될 수 있다. 유전자간의 조절 관계를 찾기 위해서는 DNA microarray나 RNAseq로부터 얻어진 시계열 전사체 정보(time series transcriptome data)를 주로 이용한다(그림 2).
그림 2. 유전자조절 네트워크(gene regulatory network) 모델링 과정
대표적인 유전자조절네트워크 모델링 방법으로는 regression-based network approach를 이용하여 유전자 조절 네트워크를 구성하는 방법 [2]과 통계학적 학습 방법과 복잡계 시뮬레이션 방법을 이용한 모델링방법[3]이 있다.
5. 조절 및 대사 네트워크 통합(integrated regulatory and metabolic network) 모델링
생명체내의 대사 네트워크와 유전자조절 네트워크는 긴밀하고 복잡하게 연결되어 있기 때문에, 유기체의 시스템 거동을 정확히 분석하고 예측하기 위해서는 대사 네트워크와 조절 네트워크 모델이 통합되어야 한다(그림 3).
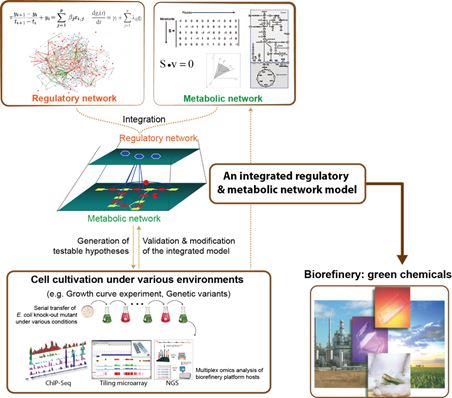
그림 3. 유전자조절 및 대사 네트워크 통합 모델 개발
하지만 유전자 발현은 전사인자(transcription factor)의 활성화부터 해당 유전자서열이 mRNA로의 발현으로 이어지는 데 수 분(minute)이 걸리는 데 반해, 대사반응은 효소의 빠른 turnover로 인해 sub-second 단위에서 이루어진다. 이렇듯 상이한 time scale과 이질적인 반응특성을 반영하여 여러 통합모델기법이 제시되고 있다[4].
통합분석의 초기모델인 regulatory flux balance analysis (rFBA)는 대사과정의 활성화 여부를 관련된 전사조절인자의 발현여부(on/off)에 의해 결정한다[5]. 하지만, rFBA와 같이 조절네트워크를 boolean logic으로 표현한 통합모델은 transcriptional regulation을 on/off로 지나치게 단순화하였다는 비판을 받고 있다. 2010년에 제시된 probabilistic regulation of metabolism (PROM)은 전사인자의 발현 정도를 확률적으로 표현하여 대사네트워크에 통합하였다[6]. 예를 들어, 전사조절인자 B가 꺼져있는 상태에서 대사관련 유전자 A가 발현될 확률은 P(A=ON|B=OFF), 전사인자 B가 켜져 있는 상태에서 유전자 A가 발현될 확률은 P(A=ON|B=ON)로 표현될 수 있으며, 이러한 조건부 확률(conditional probability)은 전사체 데이터로부터 계산된다. 이 외에도 transcriptional controlled FBA (tFBA),steady state regulatory FBA (SR-FBA) 등 여러 통합모델이 제시되고 있으나, 난해한 모델링과정과 낮은 예측도로 인해 조절 및 대사네트워크의 통합모델 개발 연구는 여전히 시스템생물학의 핵심과제이면서 풀기 어려운 문제로 남아있다.
6. 결론
상기에서 소개한 다양한 모델링 기법은 실제 생물학 연구에 적용되고 있으며, 그 중, 본 저자가 수행한 관련 연구를 예로 들어 설명한다. 학술적, 산업적으로 널리 이용되는 E. coli B 균주E(. coli BL21(DE3), E. coli REL606)의 유전체,전사체, 단백체, 형질체 등 각종 생체정보(오믹스)를 확보하고, 컴퓨터 모델링을 통해 대사네트워크를 모델링하고, 이를 이용하여 균주의 특성을 분석하였다[7]. 메탄생성 고세균 (Methanococcus maripaludis )의 전체 유전자 조절 네트워크 모델링을 통해 메탄생성경로의 조절 기작을 설명하였다[8]. 정확한 대사네트워크와 조절네트워크 구성을 위해서는 상세한 유전체 주석 정보가 우선적으로 확보되어야 한다[9, 10].
생체 네트워크를 컴퓨터 상에서 미리 모델링하고 시뮬레이션함으로써 최적의 생물시스템을 디자인한다는 개념은 기존 생물공학에서는 거의 활용되지 않았다. 하지만, 근래 들어 유전체 서열 해독기술과 유전자 합성기술이 급속히 발전하고, 다양한 분자조절 기구가 개발됨에 따라 점점 그 활용 가능성이 증대되고 있다. 지금까지의 대사공학(metabolic engineering)은 주로 연구자의 직관에 의한 시행착오 방식으로 균주를 제작하였기 때문에, 많은 경우 생산균주의 생장능력저하, 부산물의 과량생산 등의 부작용이 동반하게 되고, 공정개발에 많은 비용과 시간이 소요된다. 대량 생물정보생산 및 분석을 통해 유전자 조절 및 대사 네트워크 모델을 개발하고, 이를 이용한 컴퓨터 시뮬레이션을 통해 생명체의생리와 대사에 대한 체계적인 이해가 가능하다. 또한 이러한 가상세포 기술은 맞춤형 유전체 설계를 통한 최적의 발효 및 생산 전략을 수립하는 데 매우 유용하게 이용될 수 있다.
참고문헌
1. Price ND, Reed JL, Palsson BO: Genome-scale models of microbial cells: evaluating the consequences of constraints. Nat Rev Microbiol 2004, 2:886-897.
2. Kim H, Lee JK, Park T: Inference of large-scale gene regulatory networks using regression-based network approach. J Bioinform Comput Biol 2009, 7:717-735.
3. Bonneau R, Reiss DJ, Shannon P, Facciotti M, Hood L, Baliga NS, Thorsson V: The Inferelator: an algorithm for learning parsimonious regulatory networks from systems-biology data sets de novo. Genome Biol 2006, 7:R36.
4. Imam S, Schauble S, Brooks AN, Baliga NS, Price ND: Data-driven integration of genome-scale regulatory and metabolicnetwork models. Front Microbiol 2015, 6:409.
5. Covert MW, Knight EM, Reed JL, Herrgard MJ, Palsson BO: Integrating high-throughput and computational data elucidates bacterial networks. Nature 2004, 429:92-96.
6. Chandrasekaran S, Price ND: Probabilistic integrative modeling of genome-scale metabolic and regulatory networks in Escherichia coli and Mycobacterium tuberculosis. Proc Natl Acad Sci USA 2010, 107:17845-17850.
7. Yoon SH, Han MJ, Jeong H, Lee CH, Xia XX, Lee DH, Shim JH, Lee SY, Oh TK, Kim JF: Comparative multi-omics systems analysis of Escherichia coli strains B and K-12. Genome Biol 2012, 13:R37.
8. Yoon SH, Turkarslan S, Reiss DJ, Pan M, Burn JA, Costa KC, Lie TJ, Slagel J, Moritz RL, Hackett M, et al: A systems level predictive model for global gene regulation of methanogenesis in a hydrogenotrophic methanogen. Genome Res 2013, 23:1839-1851.
9. Kim S, Jeong H, Kim EY, Kim JF, Lee SY, Yoon SH: Genomic and transcriptomic landscape of Escherichia coli BL21(DE3).Nucleic Acids Res 2017, 45:5285-5293.
10. Yoon SH, Reiss DJ, Bare JC, Tenenbaum D, Pan M, Slagel J, Moritz RL, Lim S, Hackett M, Menon AL, et al: Parallel evolution of transcriptome architecture during genome reorganization. Genome Res 2011, 21:1892-1904.
 JOIN
JOIN LOGIN
LOGIN
 Latest update
Latest update